
Sábado, Junio 26, 2017
Los científicos de Salk mejoraron un enfoque clásico para mapear las interacciones entre proteínas
Los científicos de Salk mejoraron un enfoque clásico para mapear las interacciones entre proteínas
LA JOLLA—Los científicos de Salk han desarrollado una nueva técnica de alto rendimiento para determinar qué proteínas en una célula interactúan entre sí. El mapeo de esta red de interacciones, o "interactoma", ha sido lento en el pasado porque la cantidad de interacciones que se podían probar a la vez era limitada. El nuevo enfoque, publicado el 26 de junio en Nature Methods, permite a los investigadores probar millones de relaciones entre miles de proteínas en un solo experimento.
Haga clic aquí para una imagen de alta resolución
Crédito: Instituto Salk
"El poder de este nuevo enfoque está en la capacidad que ahora tenemos para ampliarlo", dice el autor principal. José Ecker, profesor y director del Laboratorio de Análisis Genómico de Salk e investigador del Instituto Médico Howard Hughes. "Este ensayo tiene el potencial de comenzar a abordar preguntas sobre interacciones biológicas fundamentales que no hemos podido abordar antes".
El interactoma de una célula, como un mapa de redes sociales, permite a los científicos ver quién trabaja con quién en el mundo de las proteínas. Esto les ayuda a descubrir las funciones de las diferentes proteínas y unir a los diferentes actores en las vías y procesos moleculares. Si una proteína recién descubierta interactúa con muchas otras proteínas involucradas en el metabolismo celular, por ejemplo, los investigadores pueden deducir que ese es un papel probable para la nueva proteína y potencialmente orientarla para tratamientos relacionados con la disfunción metabólica.
Hasta ahora, los investigadores generalmente se han basado en ensayos estándar de dos híbridos de levadura de alto rendimiento (Y2H) para determinar las interacciones entre las proteínas. El sistema requiere el uso de una sola proteína conocida, conocida como "cebo", para detectar un grupo de proteínas "presa". Pero encontrar todas las interacciones entre, por ejemplo, 1,000 proteínas, requeriría 1000 experimentos separados para detectar una vez a los socios de interacción de cada cebo.
"Las tecnologías actuales esencialmente requieren que las interacciones detectadas en la evaluación primaria se vuelvan a probar individualmente", dice Shelly Trigg, becaria de investigación graduada de la NSF en el Universidad de California, San Diego, en el laboratorio de Ecker, y primer autor del nuevo artículo. “Es posible que ya no sea necesario con la profundidad de detección que logra este enfoque”.
En su nuevo método, Ecker, Trigg y sus colegas agregaron un giro al ensayo Y2H estándar para una forma mucho más efectiva de medir el interactoma. Los genes de dos proteínas, cada uno en su propio círculo de ADN, se agregan a la misma célula. Si las proteínas de interés interactúan dentro de la célula, se activa un gen llamado Cre. Cuando se enciende, Cre empalma físicamente los dos círculos individuales de ADN, emparejando así los genes de las proteínas que interactúan para que el equipo pueda encontrarlos fácilmente a través de la secuenciación. El equipo puede generar una biblioteca masiva de células de levadura, cada una de las cuales contiene diferentes pares de proteínas mediante la introducción de combinaciones aleatorias de genes en ADN circular llamados plásmidos. Cuando las células son positivas para una interacción de proteínas, los investigadores pueden usar la secuenciación genética para descubrir cuáles son las dos proteínas que interactúan, utilizando nuevas tecnologías de secuenciación de ADN de alto rendimiento similares a las utilizadas para la secuenciación del genoma humano. De esta manera, ya no están limitados a probar una proteína "cebo" a la vez, sino que pueden probar las interacciones entre todas las proteínas en una biblioteca a la vez.

Haga clic aquí para una imagen de alta resolución.
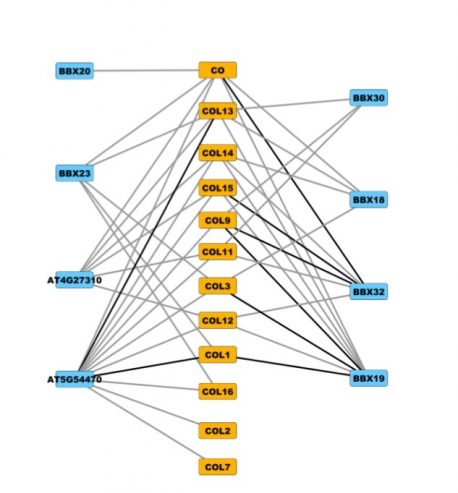
El grupo de Ecker probó el nuevo método, denominado CrY2H-seq, en todos los factores de transcripción, una gran clase de proteínas, en la planta. Arabidopsis.
“Cuando tomas 1,800 proteínas y pruebas las interacciones entre ellas, son casi 4 millones de combinaciones”, dice Ecker. “Lo hicimos diez veces en cuestión de un mes”.
Revelaron más de 8,000 interacciones entre las proteínas analizadas, lo que les dio una nueva perspectiva sobre qué Arabidopsis Los factores de transcripción interactúan entre sí. Los datos, dicen, ayudan a responder preguntas de larga data sobre si ciertos grupos de factores de transcripción tienen funciones establecidas. Descubrieron que algunos de los factores de transcripción poco conocidos interactúan con factores más conocidos que regulan la respuesta de la planta a la auxina, una hormona involucrada en la coordinación del crecimiento de la planta.
En el futuro, el método podría ampliarse para probar conjuntos más grandes de proteínas: las células humanas, por ejemplo, contienen alrededor de 20,000 XNUMX proteínas diferentes. Este método más fácil y rápido para determinar el interactoma completo de una célula también abre la posibilidad de estudiar cómo cambia el interactoma en diferentes condiciones, un experimento que nunca ha sido posible en el pasado.
Otros investigadores del estudio fueron Renee Garza, Andrew MacWilliams, Joseph Nery, Anna Bartlett, Rosa Castanon, Adeline Goubil, Joseph Feeney, Ronan O'Malley, Shao-shan Carol Huang, Zhuzhu Zhang y Mary Galli del Instituto Salk.
El trabajo y los investigadores involucrados fueron apoyados por subvenciones de la Departamento de Energía de los Estados Unidos, Programa de becas de investigación para graduados de la Fundación Nacional de Ciencias, Instituto Médico Howard Hughesy Fundación Mary K. Chapman.
PERIODICO
Nature Methods
TÍTULO
CrY2H-seq: un ensayo multiplexado masivamente para el mapeo de interactoma de cobertura profunda
AUTORES
Shelly A Trigg, Renee M Garza, Andrew MacWilliams, Joseph R Nery, Anna Bartlett, Rosa Castanon, Adeline Goubil, Joseph Feeney, Ronan O'Malley, Shao-shan C Huang, Zhuzhu Z Zhang, Mary Galli y Joseph R Ecker
PORTADA DEL DIARIO
Un método de dos híbridos de levadura multiplexada masivamente, CrY2H-seq, permite un mapeo de cobertura profunda del Arabidopsis interactoma. Portada preparada por Erin Dewalt, basada en el diseño y las ilustraciones de Shelly Trigg, Lisa Servilio y Jamie Simon en The Salk Institute for Biological Studies y por Austin Trigg en Austin Trigg Photography.
Oficina de Comunicaciones
Tel: (858) 453-4100
prensa@salk.edu
Descubrir los secretos de la vida misma es la fuerza impulsora detrás del Instituto Salk. Nuestro equipo de científicos galardonados de clase mundial traspasa los límites del conocimiento en áreas como neurociencia, investigación del cáncer, envejecimiento, inmunobiología, biología vegetal, biología computacional y más. Fundado por Jonas Salk, desarrollador de la primera vacuna contra la polio segura y eficaz, el Instituto es una organización de investigación independiente y sin fines de lucro y un hito arquitectónico: pequeño por elección, íntimo por naturaleza y valiente ante cualquier desafío.