
29 de mayo de 2012
Los científicos de Salk desarrollan una nueva técnica para resolver la estructura de los objetivos más populares para la mitad de todos los medicamentos, lo que podría ayudar al descubrimiento de nuevas terapias
Los científicos de Salk desarrollan una nueva técnica para resolver la estructura de los objetivos más populares para la mitad de todos los medicamentos, lo que podría ayudar al descubrimiento de nuevas terapias
LA JOLLA, CA—Un nuevo método para resolver rápidamente las estructuras tridimensionales de un grupo especial de proteínas, conocidas como proteínas integrales de membrana, puede acelerar el descubrimiento de fármacos al proporcionar a los científicos objetivos precisos para nuevas terapias, según un artículo publicado el 20 de mayo. en Nature Methods.
La técnica, desarrollada por científicos del Instituto Salk de Estudios Biológicos, proporciona un atajo para determinar la estructura de las proteínas integrales de membrana humana (hIMP), moléculas que se encuentran en la superficie de las células y que sirven como objetivos para aproximadamente la mitad de todos los medicamentos actuales.
Conocer la forma tridimensional exacta de los hIMP permite a los desarrolladores de fármacos comprender los mecanismos bioquímicos precisos mediante los cuales funcionan los fármacos actuales y desarrollar nuevos fármacos que se dirijan a las proteínas.
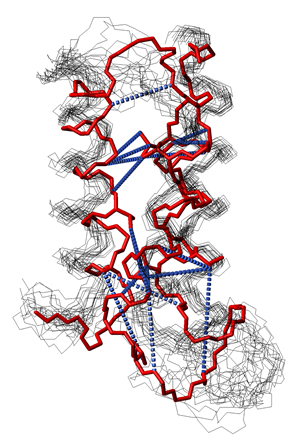
Usando su nueva técnica rápida, el equipo de Choe generó la estructura de un hIMP conocido como TMEM14A, que se muestra aquí en múltiples conformaciones tridimensionales.
Imagen: Cortesía del Instituto Salk de Estudios Biológicos
“Nuestras células contienen alrededor de 8,000 de estas proteínas, pero los biólogos estructurales han conocido la estructura tridimensional de solo 30 hIMP reportadas por todo el campo durante muchos años”, dice Senyon Choe, profesor en el Laboratorio de Biología Estructural de Salk y autor principal del artículo. “Resolvimos seis más en cuestión de meses usando esta nueva técnica. La información muy limitada sobre la forma de las proteínas de la membrana humana dificulta el diseño de fármacos basado en la estructura, pero nuestro método debería ayudar a abordar esto al aumentar drásticamente la biblioteca de estructuras hIMP conocidas”.
Las proteínas integrales de la membrana se unen a la membrana que rodea a cada célula, sirviendo como puertas de entrada para absorber nutrientes, hormonas y fármacos, eliminar productos de desecho y permitir que las células se comuniquen con su entorno. Muchas enfermedades, incluidas la enfermedad de Alzheimer, las enfermedades cardíacas y el cáncer, se han relacionado con el mal funcionamiento de los hIMP, y muchos medicamentos, que van desde la aspirina hasta los medicamentos para la esquizofrenia, se dirigen a estas proteínas.
La mayoría de los fármacos existentes se descubrieron mediante métodos de fuerza bruta que requerían examinar miles de moléculas potenciales en estudios de laboratorio para determinar si tenían un efecto terapéutico. Sin embargo, dado un modelo de la estructura 3D de un hIMP involucrado en una enfermedad específica, los desarrolladores de fármacos podrían centrarse solo en las moléculas que tienen más probabilidades de interactuar con el objetivo hIMP, ahorrando tiempo y dinero.
En el pasado, era extremadamente difícil resolver la estructura de las hIMP, debido a la dificultad de recolectarlas de las células y la dificultad de marcar los aminoácidos que componen las proteínas, un paso clave para determinar su configuración tridimensional.
"Un problema era que las hIMP cumplen muchas funciones en una célula, por lo que si intentaba diseñar células con muchas copias de las proteínas en su membrana, morirían antes de que pudiera recolectar las hIMP", dice Christian Klammt, investigador postdoctoral en Choe's. laboratorio y un primer autor del artículo.
Para evitar esto, los científicos crearon un entorno fuera de la célula, llamado sistema de expresión libre de células, para sintetizar las proteínas. Utilizaron una cámara de plexiglás que contenía todos los elementos bioquímicos necesarios para fabricar hIMP como si estuvieran dentro de la célula. Este sistema proporcionó a los investigadores suficientes proteínas para realizar un análisis estructural.

Los científicos Christian Klammt, Senyon Choe e Innokentiy Maslennikov utilizan una espectroscopia de resonancia magnética nuclear para estudios estructurales de proteínas de membrana.
Imagen: Cortesía del Instituto Salk de Estudios Biológicos
El método libre de células también les permitió agregar fácilmente aminoácidos etiquetados en el estofado bioquímico, que luego se incorporaron a las proteínas. Estos aminoácidos dieron pistas estructurales reveladoras cuando se analizaron con espectroscopía de resonancia magnética nuclear, un método para usar las propiedades magnéticas de los átomos para determinar las propiedades físicas y químicas de una molécula.
"Fue muy difícil e ineficiente introducir aminoácidos marcados de forma selectiva en la proteína producida en células vivas", dice Innokentiy Maslennikov, científico del personal de Salk y coautor del artículo. “Con un sistema libre de células, podemos controlar con precisión qué aminoácidos están disponibles para la producción de proteínas, dándonos hIMP marcados con isótopos en grandes cantidades. Usando una estrategia de etiquetado patentada, ideamos un medio para minimizar la cantidad de muestras para preparar”.
Los métodos anteriores podían tardar hasta un año en determinar la estructura de una sola proteína, pero con su nuevo método, los científicos de Salk determinaron la estructura de seis hIMP en solo 18 meses. Ya han identificado 38 hIMP más que son adecuados para el análisis con su técnica, y esperan que se use para resolver la estructura de muchos más.
Pablo Slesinger, profesor asociado en los Laboratorios de Biología de Péptidos de la Fundación Clayton de Salk, contribuyó a la investigación, al igual que científicos del Centro Conjunto de Biociencias en Corea, ETH Zurich en Suiza y la Universidad de California en San Francisco.
Otros autores del artículo fueron Monika Bayrhuber, Cédric Eichmann, Navratna Vajpai, Ellis Jeremy Chua Chiu, Katherine Blain, Luis Esquivies, June Hyun Jung Kwon, Bartosz Balana, Ursula Pieper, Andrej Sali, Witek Kwiatkowski y Roland Riek.
La investigación fue apoyada por el Programa Universitario de Clase Mundial y los Institutos Nacionales de Salud.
Sobre el Instituto Salk de Estudios Biológicos:
El Instituto Salk de Estudios Biológicos es una de las instituciones de investigación básica más importantes del mundo, donde profesores de renombre internacional investigan cuestiones fundamentales de las ciencias de la vida en un entorno único, colaborativo y creativo. Centrados tanto en el descubrimiento como en la orientación de futuras generaciones de investigadores, los científicos de Salk realizan contribuciones innovadoras a nuestra comprensión del cáncer, el envejecimiento, el Alzheimer, la diabetes y las enfermedades infecciosas mediante el estudio de la neurociencia, la genética, la biología celular y vegetal y disciplinas relacionadas.
Los logros de la facultad han sido reconocidos con numerosos honores, incluidos premios Nobel y membresías en la Academia Nacional de Ciencias. Fundado en 1960 por el pionero de la vacuna contra la polio Jonas Salk, MD, el Instituto es una organización independiente sin fines de lucro y un hito arquitectónico.
PERIODICO
Nature Methods
AUTORES
Christian Klammt, Innokentiy Maslennikov, Monika Bayrhuber, Cédric Eichmann, Navratna Vajpai, Ellis Jeremy Chua Chiu, Katherine Blain, Luis Esquivies, June Hyun Jung Kwon, Bartosz Balana, Ursula Pieper, Andrej Sali, Paul A Slesinger, Witek Kwiatkowski, Roland Riek & Senyon Choe
Oficina de Comunicaciones
Tel: (858) 453-4100
prensa@salk.edu